在Jupyter notebook中为图像添加标签,预测新图像并可视化神经网络(并使用Docker Hub共享它们!)
作者|Jenny Huang
编译|Flin
来源|towardsdatascience

作者:Jenny Huang, Ian Hunt-Isaak, William Palmer
GitHub Repo: https://github.com/ianhi/AC295-final-project-JWI
介绍
在新图像上训练图像分割模型可能会令人生畏,尤其是当你需要标记自己的数据时。为了使此任务更容易,更快捷,我们构建了一个用户友好的工具,可让你在Jupyter notebook中构建整个过程。在以下各节中,我们将向你展示我们的工具如何使你:
-
手动标记自己的图像
-
通过迁移学习建立有效的分割模型
-
可视化模型及其结果
-
以Docker映像形式共享你的项目
该工具的主要优点是易于使用,并且与现有的数据科学工作流程很好地集成在一起。通过交互式小部件和命令提示符,我们构建了一种用户友好的方式来标记图像和训练模型。
最重要的是,所有内容都可以在Jupyter notebook上运行,从而快速,轻松地建立模型,而没有太多开销。
最后,通过在Python环境中工作并使用Tensorflow和Matplotlib等标准库,可以将该工具很好地集成到现有的数据科学工作流程中,使其非常适合科学研究等用途。
例如,在微生物学中,分割细胞的显微镜图像是非常有用的。然而,随着时间的推移,跟踪细胞很可能需要分割成百上千的图像,这可能很难手动完成。在本文中,我们将使用酵母细胞的显微镜图像作为数据集,并展示我们如何构建工具来区分背景、母细胞和子细胞。
1.标签
现有许多工具可以为图像创建带标签的掩膜,包括Labelme,ImageJ甚至是图形编辑器GIMP。这些都是很棒的工具,但是它们无法集成到Jupyter notebook中,这使得它们很难与许多现有工作流程一起使用。
- Labelme:https://github.com/wkentaro/labelme
- ImageJ:https://imagej.net/Welcome
- GIMP:https://www.gimp.org/
幸运的是,Jupyter Widgets使我们能够轻松制作交互式组件并将其与我们其余的Python代码连接。
- Jupyter Widgets:https://ipywidgets.readthedocs.io/en/latest/
要在笔记本中创建训练掩膜,我们要解决两个问题:
-
用鼠标选择图像的一部分
-
轻松在图像之间切换并选择要标记的类别
为了解决第一个问题,我们使用了Matplotlib小部件后端和内置的LassoSelector。
LassoSelector会处理一条线以显示你所选择的内容,但是我们需要一些自定义代码来将掩膜绘制为覆盖层:
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.widgets import LassoSelector
from matplotlib.path import Path
class image_lasso_selector:
def __init__(self, img, mask_alpha=.75, figsize=(10,10)):
"""
img must have shape (X, Y, 3)
"""
self.img = img
self.mask_alpha = mask_alpha
plt.ioff() # see https://github.com/matplotlib/matplotlib/issues/17013
self.fig = plt.figure(figsize=figsize)
self.ax = self.fig.gca()
self.displayed = self.ax.imshow(img)
plt.ion()
lineprops = {'color': 'black', 'linewidth': 1, 'alpha': 0.8}
self.lasso = LassoSelector(self.ax, self.onselect,lineprops=lineprops, useblit=False)
self.lasso.set_visible(True)
pix_x = np.arange(self.img.shape[0])
pix_y = np.arange(self.img.shape[1])
xv, yv = np.meshgrid(pix_y,pix_x)
self.pix = np.vstack( (xv.flatten(), yv.flatten()) ).T
self.mask = np.zeros(self.img.shape[:2])
def onselect(self, verts):
self.verts = verts
p = Path(verts)
self.indices = p.contains_points(self.pix, radius=0).reshape(self.mask.shape)
self.draw_with_mask()
def draw_with_mask(self):
array = self.displayed.get_array().data
# https://en.wikipedia.org/wiki/Alpha_compositing#Straight_versus_premultiplied
self.mask[self.indices] = 1
c_overlay = self.mask[self.indices][...,None]*[1.,0,0]*self.mask_alpha
array[self.indices] = (c_overlay + self.img[self.indices]*(1-self.mask_alpha))
self.displayed.set_data(array)
self.fig.canvas.draw_idle()
def _ipython_display_(self):
display(self.fig.canvas)对于第二个问题,我们使用ipywidgets添加了漂亮的按钮和其他控件:

我们结合了这些元素(以及滚动缩放等改进功能)来制作了一个标签控制器对象。现在,我们可以拍摄酵母的显微镜图像并分割母细胞和子细胞:
套索选择图像标签演示:
https://youtu.be/aYb17GueVcU
你可以查看完整的对象,它允许你滚动,缩放,右键单击以平移,并在此处(https://github.com/ianhi/AC295-final-project-JWI/blob/2bacc09c06228c1eb49130ec5aaeff660f921033/lib/labelling.py#L152) 选择多个类。
现在我们可以在notebook上标记少量图像,将它们保存到正确的文件夹结构中,然后开始训练CNN!
2.模型训练
模型
U-net是一个卷积神经网络,最初设计用于分割生物医学图像,但已成功用于许多其他类型的图像。它以现有的卷积网络为基础,可以在很少的训练图像的情况下更好地工作,并进行更精确的分割。这是一个最新模型,使用segmentation_models库也很容易实现。
- segmentation_models库:https://github.com/qubvel/segmentation_models
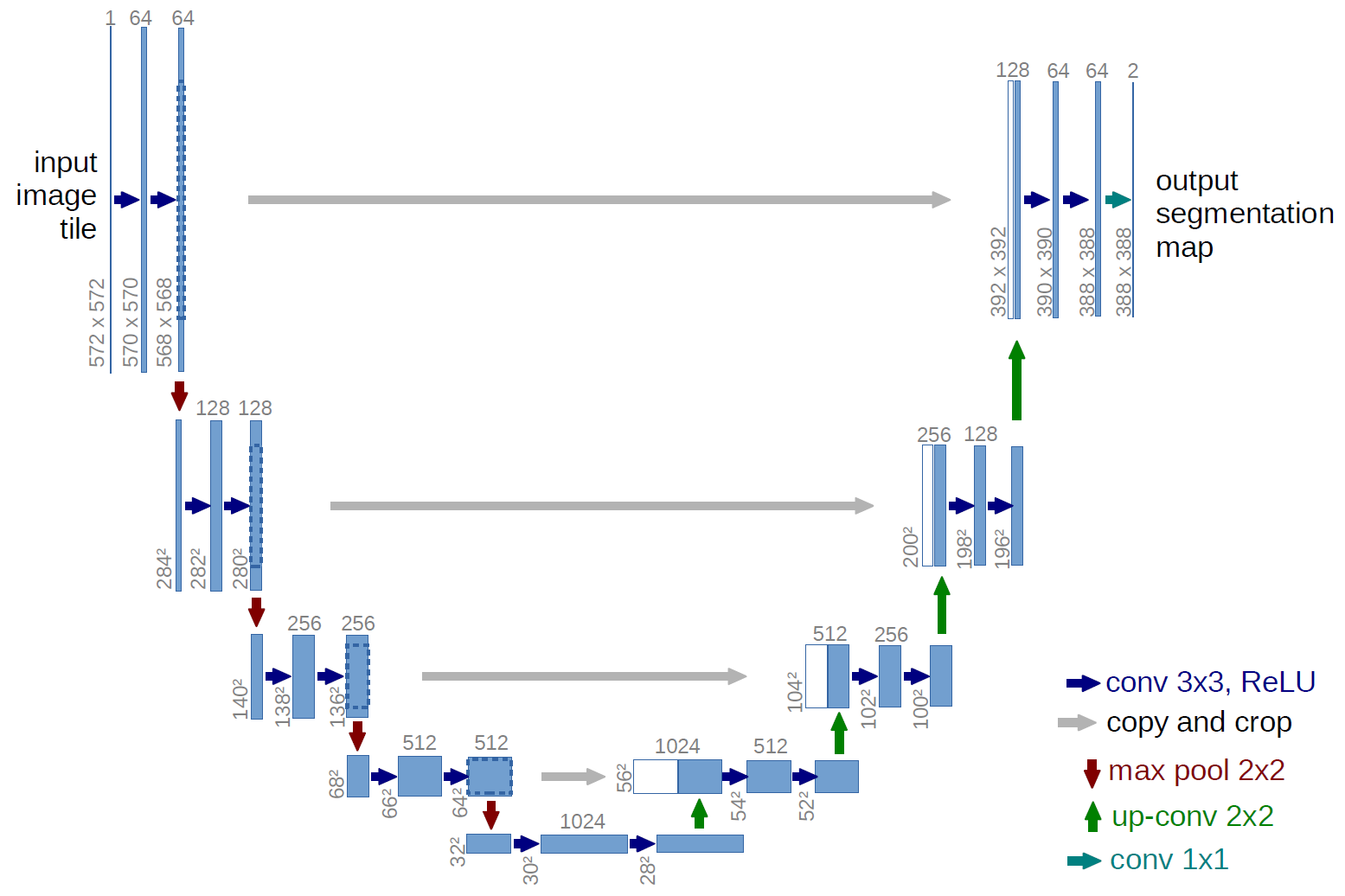
U-net的独特之处在于它通过交叉连接将编码器和解码器结合在一起(上图中的灰色箭头)。这些跳跃连接从下采样路径中的相同大小的部分跨到上采样路径。这样可以提高你对上采样时输入到模型中的原始像素的了解,这已被证明可以提高分割任务的性能。
尽管U-net很棒,但是如果我们没有给它足够的训练示例,它将无法正常工作。考虑到手动分割图像的繁琐工作,我们仅手动标记了13张图像。仅用很少的训练示例,不可能训练具有数百万个参数的神经网络。为了克服这个问题,我们既需要数据扩充又需要迁移学习。
数据扩充
自然,如果你的模型具有很多参数,则需要成比例的训练示例才能获得良好的性能。使用图像和掩膜的小型数据集,我们可以创建新图像,这些图像对于模型和原始图像一样具有洞察力和实用性。
我们该怎么做?我们可以翻转图像,旋转角度,向内或向外缩放图像,裁剪,平移图像,甚至可以通过添加噪点来模糊图像,但最重要的是,我们可以将这些操作结合起来以创建许多新的训练例子。
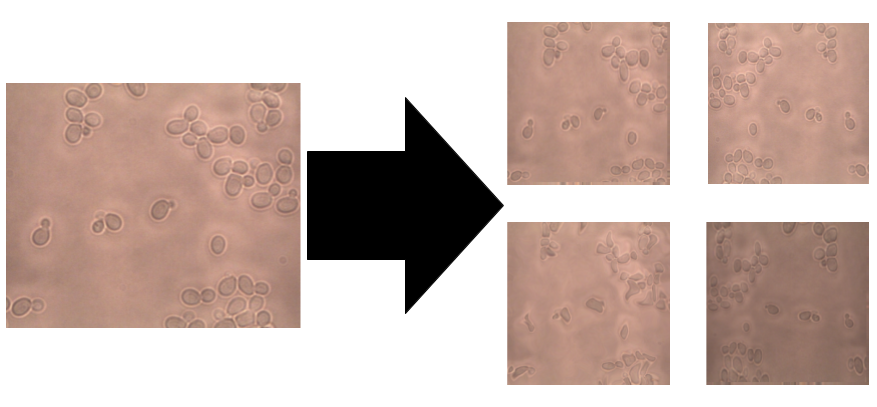
与分类相比,图像数据增强在分割方面具有更多的复杂性。对于分类,你只需要放大图像,因为标签将保持不变(0或1或2…)。但是,对于分割,还需要与图像同步转换标签(作为掩膜)。为此,我们将albumentations库与自定义数据生成器一起使用,因为据我们所知,Keras ImageDataGenerator当前不支持组合“Image+mask”。
import albumentations as A
import numpy as np
from tensorflow.keras.preprocessing.image import ImageDataGenerator
TARGET_SIZE = (224,224)
BATCH_SIZE = 6
def create_augmentation_pipeline():
augmentation_pipeline = A.Compose(
[
A.HorizontalFlip(p = 0.5), # Apply horizontal flip to 50% of images
A.OneOf(
[
# Apply one of transforms to 50% of images
A.RandomContrast(), # Apply random contrast
A.RandomGamma(), # Apply random gamma
A.RandomBrightness(), # Apply random brightness
],
p = 0.5
),
A.OneOf(
[
# Apply one of transforms to 50% images
A.ElasticTransform(
alpha = 120,
sigma = 120 * 0.05,
alpha_affine = 120 * 0.03
),
A.GridDistortion()
],
p = 0.5
)
],
p = 0.9 # In 10% of cases keep same image because that's interesting also
)
return augmentation_pipeline
def create_datagenerator(PATH):
options = {'horizontal_flip': True, 'vertical_flip': True}
image_datagen = ImageDataGenerator(rescale=1./255, **options)
mask_datagen = ImageDataGenerator(**options)
val_datagen = ImageDataGenerator(rescale=1./255)
val_datagen_mask = ImageDataGenerator(rescale=1)
# Create custom zip and custom batch_size
def combine_generator(gen1, gen2, batch_size=6,training=True):
while True:
image_batch, label_batch = next(gen1)[0], np.expand_dims(next(gen2)[0][:,:,0],axis=-1)
image_batch, label_batch = np.expand_dims(image_batch,axis=0), np.expand_dims(label_batch,axis=0)
for i in range(batch_size - 1):
image_i,label_i = next(gen1)[0], np.expand_dims(next(gen2)[0][:,:,0],axis=-1)
if training == True:
aug_pipeline = create_augmentation_pipeline()
augmented = aug_pipeline(image=image_i, mask=label_i)
image_i, label_i = augmented['image'], augmented['mask']
image_i, label_i = np.expand_dims(image_i,axis=0),np.expand_dims(label_i,axis=0)
image_batch = np.concatenate([image_batch,image_i],axis=0)
label_batch = np.concatenate([label_batch,label_i],axis=0)
yield((image_batch,label_batch))
seed = np.random.randint(0,1e5)
train_image_generator = image_datagen.flow_from_directory(PATH+'train_imgs', seed=seed, target_size=TARGET_SIZE, class_mode=None, batch_size=BATCH_SIZE)
train_mask_generator = mask_datagen.flow_from_directory(PATH+'train_masks', seed=seed, target_size=TARGET_SIZE, class_mode=None, batch_size=BATCH_SIZE)
train_generator = combine_generator(train_image_generator, train_mask_generator,training=True)
val_image_generator = val_datagen.flow_from_directory(PATH+'val_imgs', seed=seed, target_size=TARGET_SIZE, class_mode=None, batch_size=BATCH_SIZE)
val_mask_generator = val_datagen_mask.flow_from_directory(PATH+'val_masks', seed=seed, target_size=TARGET_SIZE, class_mode=None, batch_size=BATCH_SIZE)
val_generator = combine_generator(val_image_generator, val_mask_generator,training=False)
return train_generator, val_generator迁移学习
即使我们现在已经创建了100个或更多的图像,但这仍然不够,因为U-net模型具有超过600万个参数。这也是迁移学习发挥作用的地方。
迁移学习可以让你在一个任务上训练一个模型,并将其重用于另一项类似任务。它极大地减少了你的训练时间,更重要的是,即使像我们这样的小型训练,它也可以产生有效的模型。例如,诸如MobileNet,Inception和DeepNet之类的神经网络通过训练大量图像来学习特征空间,形状,颜色,纹理等。然后,我们可以通过获取这些模型权重并对其进行稍微修改以激活我们自己的训练图像中的模式来传递所学的内容。
现在我们如何使用U-net的迁移学习呢?我们使用segmentation_models库来实现这一点。我们使用你选择的深层神经网络(MobileNet、Inception、ResNet)的各层以及在图像分类(ImageNet)上找到的参数,并将它们用作U-net的前半部分(编码器)。然后,使用自己的扩展数据集训练解码器层。
整理
我们将所有这些放到了Segmentation模型类中,你可以在此处找到。
创建模型对象时,会出现一个交互式命令提示符,你可以在其中自定义U-net的各个方面,例如损失函数,骨干网等:
经过30个星期的训练,我们达到了95%的准确性。请注意,选择良好的损失函数很重要。我们首先尝试了交叉熵损失,但是该模型无法区分相貌相似的母细胞和子细胞,并且由于看到的非酵母像素多于酵母像素的类不平衡,因此该模型的性能不佳。
我们发现使用Dice loss可以获得更好的结果。Dice loss与IOU相关联,通常更适合分割任务,因为它可以缩小预测值和真实值之间的差距。

3.可视化
现在我们的模型已经训练完毕,让我们使用一些可视化技术来查看其工作原理。
我们按照Ankit Paliwal的教程进行操作。你可以在他相应的GitHub存储库中找到实现。在本节中,我们将在酵母细胞分割模型上可视化他的两种技术,即中间层激活和类激活的热图。
- 参考教程:https://towardsdatascience.com/understanding-your-convolution-network-with-visualizations-a4883441533b
- GitHub存储库:https://github.com/anktplwl91/visualizing_convnets
中间层激活
第一种技术在测试图像上显示网络前向传播时中间层的输出。这使我们可以看到输入图像的哪些特征在每一层都突出显示。输入测试图像后,我们将网络中一些卷积层的前几个输出可视化:
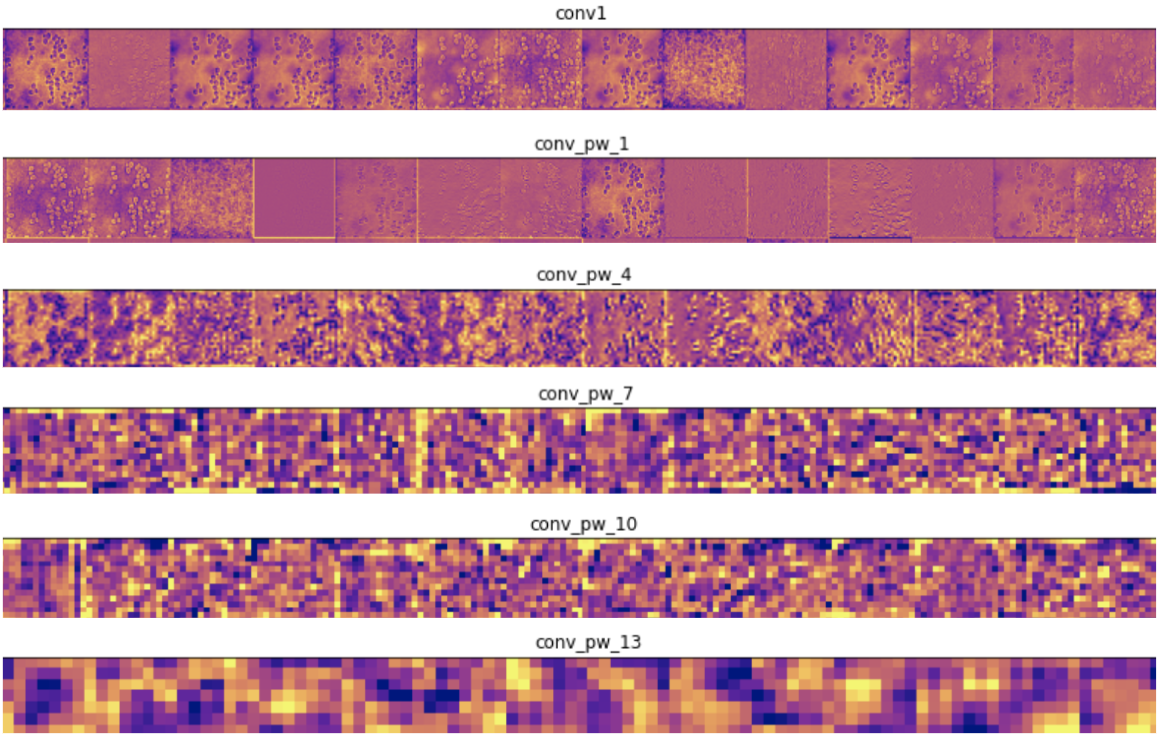
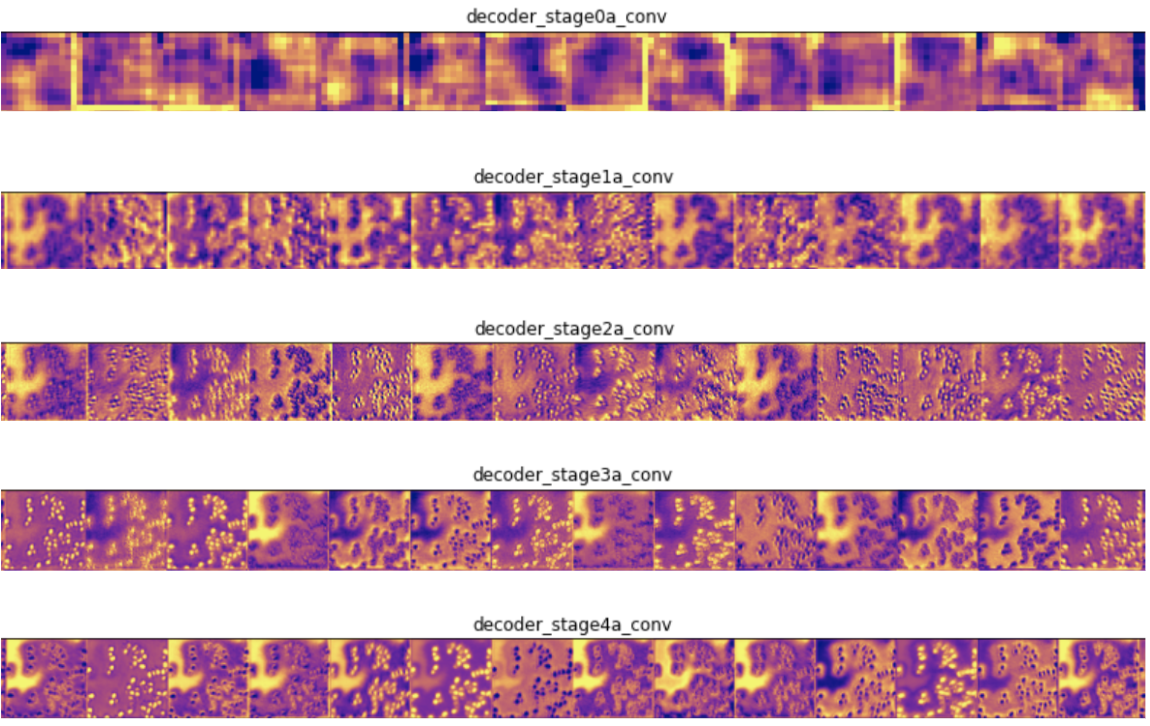
在编码器层中,靠近输入的过滤器可检测更多细节,而靠近模型输出的过滤器可检测更一般的特征,这是意料之中的。在解码器层中,我们看到了相反的模式,即从抽象到更具体的细节,这也是意料之中的。
类激活的热图
接下来,我们看一下类激活图。这些热图让你了解图像的每个位置对于预测输出类别的重要性。在这里,我们将酵母细胞模型的最后一层可视化,因为类预测标签在很大程度上依赖于它。
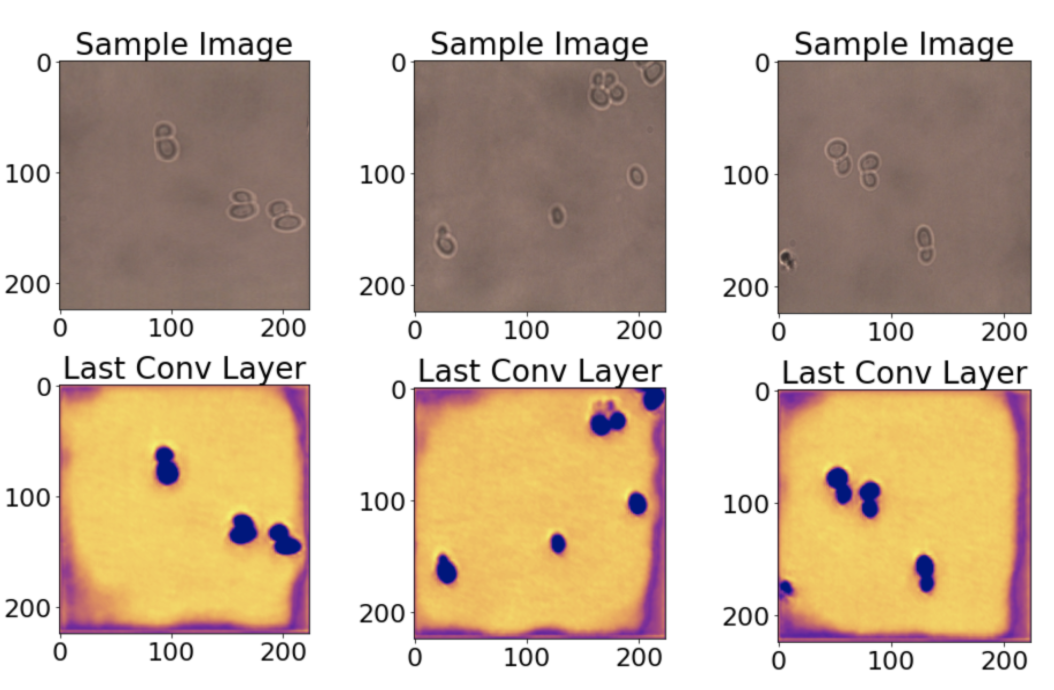
从热图可以看出,细胞位置以及部分图像边界已被正确激活,这有些令人惊讶。
我们还研究了本教程中的最后一项技术,该技术显示了每个卷积滤波器最大程度地响应哪些图像,但是对于我们特定的酵母细胞模型,可视化效果并不是很有用。
4.制作和共享Docker映像
找到很棒的模型并尝试运行它,却发现由于神秘的依赖关系问题,它在你的环境中不起作用,这非常令人沮丧。
我们通过为我们的工具创建一个Docker镜像来解决这个问题。这使我们可以完全定义运行代码的环境,甚至是操作系统。
对于此项目,我们基于Jupyter Docker Stacks的jupyter/tensorflow-notebook镜像建立Docker镜像。然后,我们仅添加了几行代码来安装所需的库,并将GitHub存储库的内容复制到Docker映像中。
如果你好奇,可以在此处查看我们最终的Dockerfile 。最后,我们将此映像推送到Docker Hub。你可以通过运行以下命令进行尝试:
sudo docker run -p 8888:8888 ianhuntisaak/ac295-final-project:v3 \
-e JUPYTER_LAB_ENABLE=yes结论与未来工作
使用此工具,你可以以用户友好的方式轻松地在新图像上训练分割模型。虽然有效,但在可用性,自定义和模型性能方面仍有改进的空间。将来,我们希望:
- 通过使用html5画布构建自定义Jupyter小部件来改善套索工具,以减少手动分割时的滞后
- 探索新的损失函数和模型作为迁移学习的基础
- 使解释可视化更加容易,并向用户建议改善结果的方法
欢迎关注磐创AI博客站:
http://panchuang.net/
sklearn机器学习中文官方文档:
http://sklearn123.com/
欢迎关注磐创博客资源汇总站:
http://docs.panchuang.net/
原创文章,作者:磐石,如若转载,请注明出处:https://panchuang.net/2020/10/19/%e9%80%9a%e8%bf%87%e8%bf%81%e7%a7%bb%e5%ad%a6%e4%b9%a0%e5%bb%ba%e7%ab%8b%e4%b8%80%e4%b8%aa%e6%98%93%e4%ba%8e%e4%bd%bf%e7%94%a8%e7%9a%84%e5%9b%be%e5%83%8f%e5%88%86%e5%89%b2%e5%b7%a5%e5%85%b7/

