数字病理学是随着患者组织样本的数字化,特别是数字整张幻灯片图像(WSIS)的使用而出现的。全玻片成像(WSI)是指对常规玻片进行扫描以产生数字幻灯片,是世界范围内病理科采用的最新的成像方式。
由于WSI具有非常大的维度,因此以原始大小分析它们会产生两个问题:
为什么选择基于补丁的方法?
直接使用CNN进行WSI分类有几个缺点。首先,需要大量的图像下采样,这样可能会丢失大部分可区分的细节。其次,CNN可能只从图像中的多个区分模式中的一个模式中学习,从而导致数据效率低下。区分信息被编码在高分辨率图像块中。因此,一种解决方案是在高分辨率图像补丁上训练CNN,并基于补丁级别的预测来预测WSI的标签。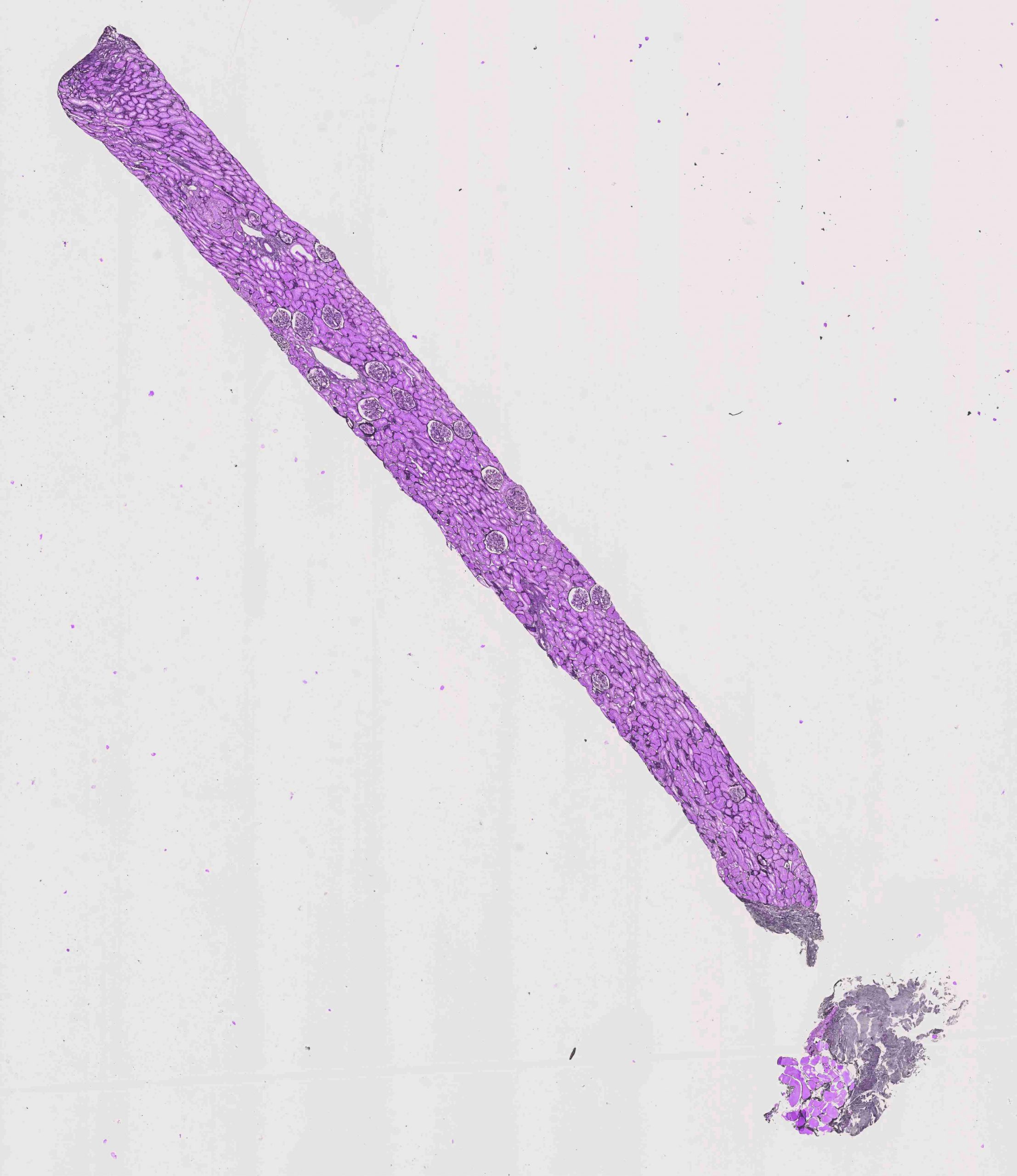
上图的形状为16000 X 18500,并以‘TIFF’格式存储,每个肾小球的注释也可以在‘xml’文件中找到,该文件具有肾小球的类型(正常或异常)及其坐标(作为多边形)。
我使用了OpenSlide库,它允许我们以不同的放大倍数阅读WSI。但是,由于库的依赖关系,它们在导入库时可能会遇到挑战。OpenSlide
import numpy as np
import matplotlib.pyplot as plt
import cv2 as cv
import os
import openslide
import xml.etree.cElementTree as ET
import random
import os
import glob定义要读取WSI的放大级别。当mag_level=0时,这意味着您在没有任何下采样的情况下读取原始级别的WSI。
mag_level = 2 # here I am reading the WSI at level 2
factor = 2**mag_level WSI文件夹和批注文件夹的路径及其名称:
slidepath = "F:/Renal_Vasculitis/Data/WSI" #path to folder of WSIs
annotpath = "F:/Renal_Vasculitis/Data/Annotations"
slidename = "EE 833604 nr17clean.tiff"
annotname = "EE 833604 nr17clean.xml"我们通过解析xml文件,读取标记
您可以通过以下方式获取WSI中存在的批注列表:
annolist = parse_xml(os.path.join(annotpath,annotname))仅使用上面的注释列表提取补丁,该列表丢弃了WSI的背景和不重要部分:
第3-4行:使用OpenSlide读取WSI并获取作为年鉴的注释列表
第5-7行:对于每个肾小球,即Annolist[i],将坐标作为坐标,并生成一个返回左上角(x和y)的边界框。我们将使用这个x和y来读取WSI中的部分。
第8-11行:此for循环通过随机偏移原始x和y,即spointx,spointy,为每个肾小球生成30个补丁。k的取值范围由用户根据需要决定,可以改变偏移量的取值范围,以获得同一肾小球的更多/更少的补丁。
第12行:在这里,我们乘以因子,得到WSI中的原始坐标,而不是按照级别。(因为年鉴中的坐标是根据高程,而不是原始的WSI坐标)
第13&14行:使用OpenSlide中接受输入的read_region函数:
i.左上角(spointx0,spointy0)
二、二、读取WSI的放大级别
三、三、面片大小(例如,256×256)
第13行:使用cv.imwrite存储图像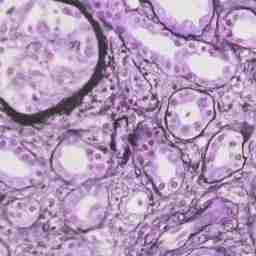
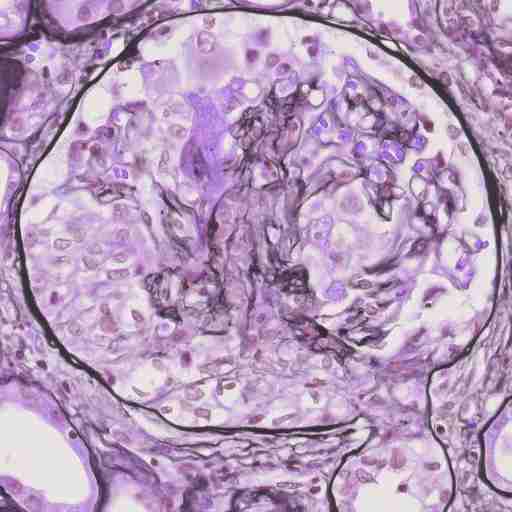
编码愉快!
原创文章,作者:fendouai,如若转载,请注明出处:https://panchuang.net/2021/07/11/%e4%bb%8e%e6%95%b4%e4%b8%aa%e5%b9%bb%e7%81%af%e7%89%87%e5%9b%be%e5%83%8f%e4%b8%ad%e6%8f%90%e5%8f%96%e8%a1%a5%e4%b8%81wsi-2/

