医学图像 | 使用深度学习实现乳腺癌分类(附python演练)

来源|Towards Data Science
当乳腺细胞生长失控时,乳腺癌就开始了。这些细胞通常形成一个肿瘤,通常可以在x光片上直接看到或感觉到有一个肿块。如果癌细胞能生长到周围组织或扩散到身体的其他地方,那么这个肿瘤就是恶性的。
以下是报告:
-
大约八分之一的美国女性(约12%)将在其一生中患上浸润性乳腺癌。
-
2019年,美国预计将有268,600例新的侵袭性乳腺癌病例,以及62,930例新的非侵袭性乳腺癌。
-
大约85%的乳腺癌发生在没有乳腺癌家族史的女性身上。这些发生是由于基因突变,而不是遗传突变
-
如果一名女性的一级亲属(母亲、姐妹、女儿)被诊断出患有乳腺癌,那么她患乳腺癌的风险几乎会增加一倍。在患乳腺癌的女性中,只有不到15%的人的家人被诊断出患有乳腺癌。
挑战
构建一个算法,通过查看活检图像自动识别患者是否患有乳腺癌。算法必须非常精确,因为人的生命安全是第一的。
数据
数据集可以从这里(https://web.inf.ufpr.br/vri/databases/breast-cancer-histopathological-database-breakhis/)下载。这是二分类问题。我把数据拆分如图所示
dataset train
benign
b1.jpg
b2.jpg
//
malignant
m1.jpg
m2.jpg
// validation
benign
b1.jpg
b2.jpg
//
malignant
m1.jpg
m2.jpg
//...
训练文件夹在每个类别中有1000个图像,而验证文件夹在每个类别中有250个图像。
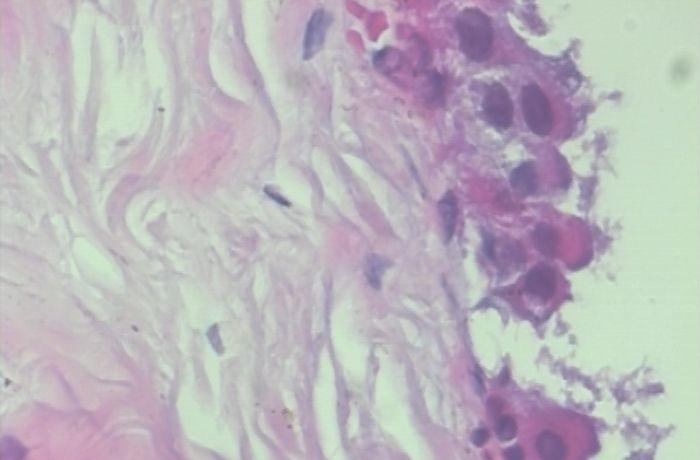
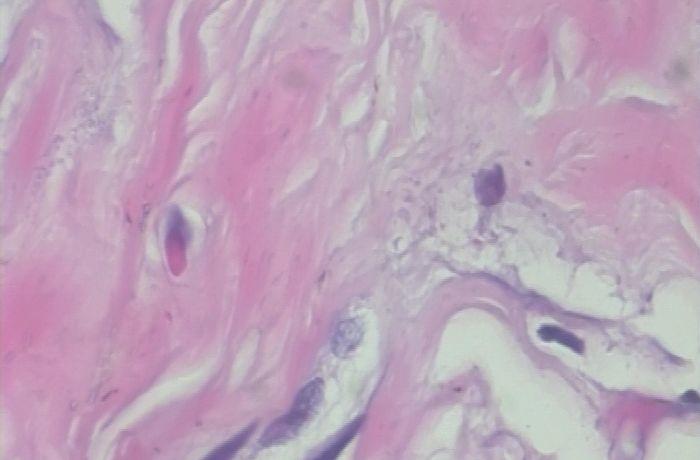
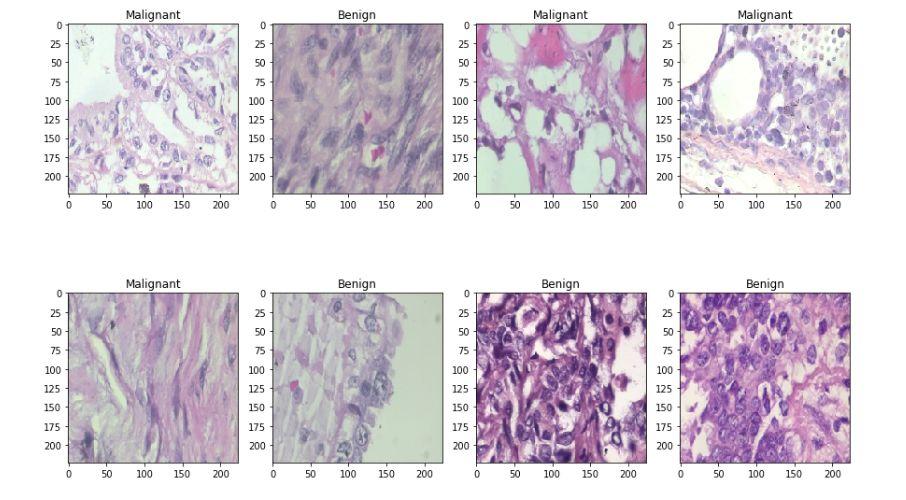
以上两张图片是良性样本
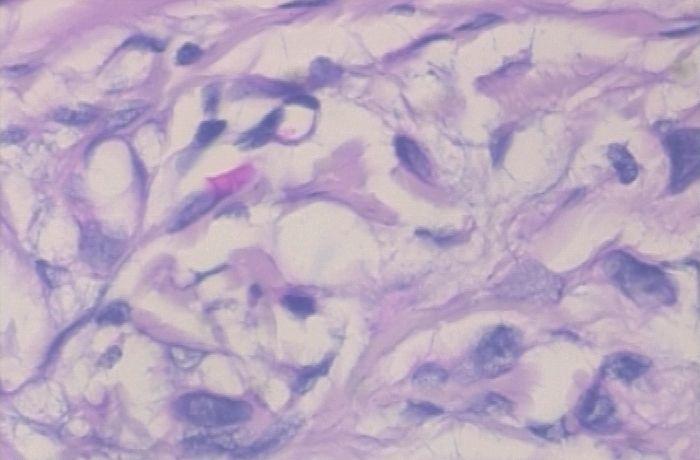
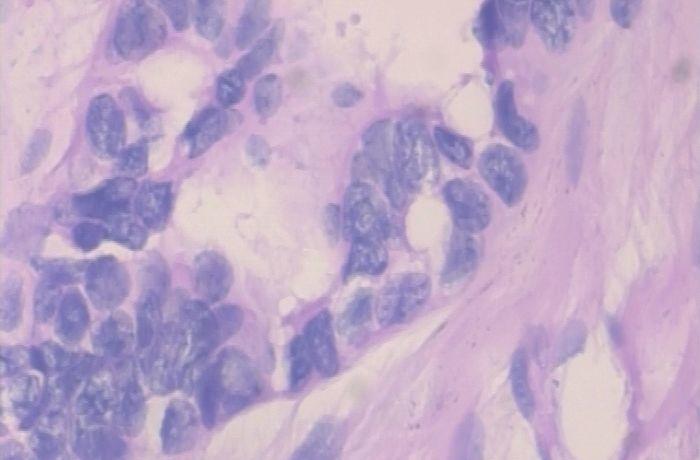
以上两张图片是恶性样本
环境和工具
-
scikit-learn
-
keras
-
numpy
-
pandas
-
matplotlib
图像分类
完整的图像分类流程可以形式化如下:
我们的输入是一个由N个图像组成的训练数据集,每个图像都有相应的标签。
然后,我们使用这个训练集来训练分类器,来学习每个类。
最后,我们通过让分类器预测一组从未见过的新图像的标签来评估分类器的质量。然后我们将这些图像的真实标签与分类器预测的标签进行比较。
代码实现
让我们开始使用代码。github上的完整项目可以在此链接(https://github.com/abhinavsagar/Breast-cancer-classification)。
让我们从加载所有库和依赖项开始。
import json
import math
import os
import cv2
from PIL import Image
import numpy as np
from keras import layers
from keras.applications import DenseNet201
from keras.callbacks import Callback, ModelCheckpoint, ReduceLROnPlateau, TensorBoard
from keras.preprocessing.image import ImageDataGenerator
from keras.utils.np_utils import to_categorical
from keras.models import Sequential
from keras.optimizers import Adam
import matplotlib.pyplot as plt
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.metrics import cohen_kappa_score, accuracy_score
import scipy
from tqdm import tqdm
import tensorflow as tf
from keras import backend as K
import gc
from functools import partial
from sklearn import metrics
from collections import Counter
import json
import itertools
接下来,我将图像加载到相应的文件夹中。
def Dataset_loader(DIR, RESIZE, sigmaX=10):
IMG = []
read = lambda imname: np.asarray(Image.open(imname).convert("RGB"))
for IMAGE_NAME in tqdm(os.listdir(DIR)):
PATH = os.path.join(DIR,IMAGE_NAME)
_, ftype = os.path.splitext(PATH)
if ftype == ".png":
img = read(PATH)
img = cv2.resize(img, (RESIZE,RESIZE))
IMG.append(np.array(img))
return IMG
benign_train = np.array(Dataset_loader('data/train/benign',224))
malign_train = np.array(Dataset_loader('data/train/malignant',224))
benign_test = np.array(Dataset_loader('data/validation/benign',224))
malign_test = np.array(Dataset_loader('data/validation/malignant',224))
之后,我创建了一个全0的numpy数组,用于标记良性图像,以及全1的numpy数组,用于标记恶性图像。我还重新整理了数据集,并将标签转换为分类格式。
benign_train_label = np.zeros(len(benign_train))
malign_train_label = np.ones(len(malign_train))
benign_test_label = np.zeros(len(benign_test))
malign_test_label = np.ones(len(malign_test))
X_train = np.concatenate((benign_train, malign_train), axis = 0)
Y_train = np.concatenate((benign_train_label, malign_train_label), axis = 0)
X_test = np.concatenate((benign_test, malign_test), axis = 0)
Y_test = np.concatenate((benign_test_label, malign_test_label), axis = 0)
s = np.arange(X_train.shape[0])
np.random.shuffle(s)
X_train = X_train[s]
Y_train = Y_train[s]
s = np.arange(X_test.shape[0])
np.random.shuffle(s)
X_test = X_test[s]
Y_test = Y_test[s]
Y_train = to_categorical(Y_train, num_classes= 2)
Y_test = to_categorical(Y_test, num_classes= 2)
然后我将数据集分成两组,分别具有80%和20%图像的训练集和测试集。让我们看一些样本良性和恶性图像。
x_train, x_val, y_train, y_val = train_test_split(
X_train, Y_train,
test_size=0.2,
random_state=11
)
w=60
h=40
fig=plt.figure(figsize=(15, 15))
columns = 4
rows = 3
for i in range(1, columns*rows +1):
ax = fig.add_subplot(rows, columns, i)
if np.argmax(Y_train[i]) == 0:
ax.title.set_text('Benign')
else:
ax.title.set_text('Malignant')
plt.imshow(x_train[i], interpolation='nearest')
plt.show()
我使用的batch值为16。batch是深度学习中最重要的超参数之一。我更喜欢使用更大的batch来训练我的模型,因为它允许从gpu的并行性中提高计算速度。但是,众所周知,batch太大会导致泛化效果不好。在一个极端下,使用一个等于整个数据集的batch将保证收敛到目标函数的全局最优。但是这是以收敛到最优值较慢为代价的。另一方面,使用更小的batch已被证明能够更快的收敛到好的结果。这可以直观地解释为,较小的batch允许模型在必须查看所有数据之前就开始学习。使用较小的batch的缺点是不能保证模型收敛到全局最优。因此,通常建议从小batch开始,通过训练慢慢增加batch大小来加快收敛速度。
我还做了一些数据扩充。数据扩充的实践是增加训练集规模的一种有效方式。训练实例的扩充使网络在训练过程中可以看到更加多样化,仍然具有代表性的数据点。
然后,我创建了一个数据生成器,自动从文件夹中获取数据。Keras为此提供了方便的python生成器函数。
BATCH_SIZE = 16
train_generator = ImageDataGenerator(
zoom_range=2, # 设置范围为随机缩放
rotation_range = 90,
horizontal_flip=True, # 随机翻转图片
vertical_flip=True, # 随机翻转图片
)
下一步是构建模型。这可以通过以下3个步骤来描述:
-
我使用DenseNet201作为训练前的权重,它已经在Imagenet比赛中训练过了。设置学习率为0.0001。
-
在此基础上,我使用了globalaveragepooling层和50%的dropout来减少过拟合。
-
我使用batch标准化和一个以softmax为激活函数的含有2个神经元的全连接层,用于2个输出类的良恶性。
-
我使用Adam作为优化器,使用二元交叉熵作为损失函数。
def build_model(backbone, lr=1e-4):
model = Sequential()
model.add(backbone)
model.add(layers.GlobalAveragePooling2D())
model.add(layers.Dropout(0.5))
model.add(layers.BatchNormalization())
model.add(layers.Dense(2, activation='softmax'))
model.compile(
loss='binary_crossentropy',
optimizer=Adam(lr=lr),
metrics=['accuracy']
)
return model
resnet = DenseNet201(
weights='imagenet',
include_top=False,
input_shape=(224,224,3)
)
model = build_model(resnet ,lr = 1e-4)
model.summary()
让我们看看每个层中的输出形状和参数。

在训练模型之前,定义一个或多个回调函数很有用。非常方便的是:ModelCheckpoint和ReduceLROnPlateau。
-
ModelCheckpoint:当训练通常需要多次迭代并且需要大量的时间来达到一个好的结果时,在这种情况下,ModelCheckpoint保存训练过程中的最佳模型。
-
ReduceLROnPlateau:当度量停止改进时,降低学习率。一旦学习停滞不前,模型通常会从将学习率降低2-10倍。这个回调函数会进行监视,如果在’patience'(耐心)次数下,模型没有任何优化的话,学习率就会降低。
该模型我训练了20个epoch。
learn_control = ReduceLROnPlateau(monitor='val_acc', patience=5,
verbose=1,factor=0.2, min_lr=1e-7)
filepath="weights.best.hdf5"
checkpoint = ModelCheckpoint(filepath, monitor='val_acc', verbose=1, save_best_only=True, mode='max')
history = model.fit_generator(
train_generator.flow(x_train, y_train, batch_size=BATCH_SIZE),
steps_per_epoch=x_train.shape[0] / BATCH_SIZE,
epochs=20,
validation_data=(x_val, y_val),
callbacks=[learn_control, checkpoint]
)
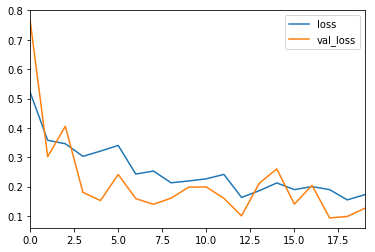
性能指标
评价模型性能最常用的指标是精度。然而,当您的数据集中只有2%属于一个类(恶性),98%属于其他类(良性)时,错误分类的分数就没有意义了。你可以有98%的准确率,但仍然没有发现恶性病例,即预测的时候全部打上良性的标签,这是一个不好的分类器。
history_df = pd.DataFrame(history.history)
history_df[['loss', 'val_loss']].plot()
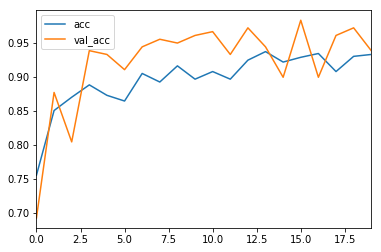
history_df = pd.DataFrame(history.history)
history_df[['acc', 'val_acc']].plot()
精度,召回率和F1度量
为了更好地理解错误分类,我们经常使用以下度量来更好地理解真正例(TP)、真负例(TN)、假正例(FP)和假负例(FN)。
精度反映了被分类器判定的正例中真正的正例样本的比重。
召回率反映了所有真正为正例的样本中被分类器判定出来为正例的比例。
F1度量是准确率和召回率的调和平均值。
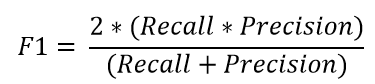
F1度量越高,模型越好。对于所有三个度量,0值表示最差,而1表示最好。
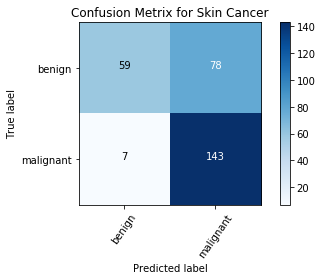
混淆矩阵
混淆矩阵是分析误分类的一个重要指标。矩阵的每一行表示预测类中的实例,而每一列表示实际类中的实例。对角线表示已正确分类的类。这很有帮助,因为我们不仅知道哪些类被错误分类,还知道它们为什么被错误分类。
from sklearn.metrics import classification_report
classification_report( np.argmax(Y_test, axis=1), np.argmax(Y_pred_tta, axis=1))
from sklearn.metrics import confusion_matrix
def plot_confusion_matrix(cm, classes,
normalize=False,
title='Confusion matrix',
cmap=plt.cm.Blues):
if normalize:
cm = cm.astype('float') / cm.sum(axis=1)[:, np.newaxis]
print("Normalized confusion matrix")
else:
print('Confusion matrix, without normalization')
print(cm)
plt.imshow(cm, interpolation='nearest', cmap=cmap)
plt.title(title)
plt.colorbar()
tick_marks = np.arange(len(classes))
plt.xticks(tick_marks, classes, rotation=55)
plt.yticks(tick_marks, classes)
fmt = '.2f' if normalize else 'd'
thresh = cm.max() / 2.
for i, j in itertools.product(range(cm.shape[0]), range(cm.shape[1])):
plt.text(j, i, format(cm[i, j], fmt),
horizontalalignment="center",
color="white" if cm[i, j] > thresh else "black")
plt.ylabel('True label')
plt.xlabel('Predicted label')
plt.tight_layout()
cm = confusion_matrix(np.argmax(Y_test, axis=1), np.argmax(Y_pred, axis=1))
cm_plot_label =['benign', 'malignant']
plot_confusion_matrix(cm, cm_plot_label, title ='Confusion Metrix for Skin Cancer')
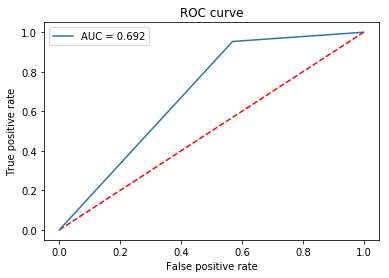
ROC曲线
45度的线代表是随机线,其中曲线下面积或AUC是0.5。该线的曲线越远,AUC越高,模型越好。模型可以获得的最高值是AUC为1,其中曲线形成直角三角形。ROC曲线还可以帮助调试模型。例如,如果曲线的左下角更接近随机线,则意味着模型在Y = 0时错误分类。然而,如果它在右上方是随机的,则意味着错误发生在Y = 1。
from sklearn.metrics import roc_auc_score, auc
from sklearn.metrics import roc_curve
roc_log = roc_auc_score(np.argmax(Y_test, axis=1), np.argmax(Y_pred_tta, axis=1))
false_positive_rate, true_positive_rate, threshold = roc_curve(np.argmax(Y_test, axis=1), np.argmax(Y_pred_tta, axis=1))
area_under_curve = auc(false_positive_rate, true_positive_rate)
plt.plot([0, 1], [0, 1], 'r--')
plt.plot(false_positive_rate, true_positive_rate, label='AUC = {:.3f}'.format(area_under_curve))
plt.xlabel('False positive rate')
plt.ylabel('True positive rate')
plt.title('ROC curve')
plt.legend(loc='best')
plt.show()
#plt.savefig(ROC_PLOT_FILE, bbox_inches='tight')
plt.close()
结果

结论
虽然这个项目还远未完成,但看到深度学习在如此多样的现实世界问题中取得成功是值得注意的。在这个博客中,我演示了如何使用卷积神经网络和迁移学习从一组显微图像中对良性和恶性乳腺癌进行分类。
你也许还想看:
● 【ECCV 2018】谷歌AI超大规模图像竞赛,中国团队获目标检测冠军
欢迎扫码关注:
原创文章,作者:fendouai,如若转载,请注明出处:https://panchuang.net/2019/11/09/95720f2f90/


 点击下方
点击下方